Informationssyssteme zur Unterstützung von Erforschung, Diagnose und Therapie angeborener Stoffwechselerkrankungen
Bei den angeborenen Stoffwechselerkrankungen [1] handelt es sich um Störungen des Gesamt- oder eines Teilstoffwechsels, die zur Bildung qualitativ oder quantitativ abnormer Stoffwechselzwischenprodukte führen und das sich daraus ergebende Krankheitsbild hervorrufen. Diese Störungen beruhen auf dem Fehlen oder dem Defekt eines Enzyms und werden auch als Enzymopathien bezeichnet. Die Ursache dafür liegt in einem Gendefekt, der an die Nachkommen vererbt wird.
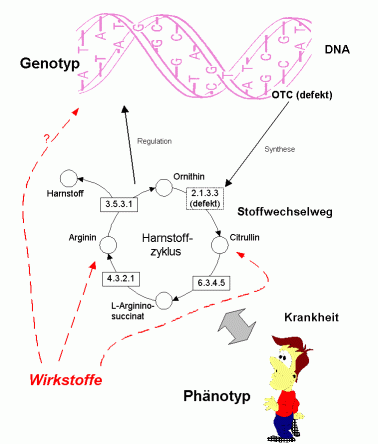
Die häufigste angeborene Stoffwechselkrankheit des Harnstoffzyklus ist der Ornithintranscarbamylase-Mangel (OTC-Mangel). Diese Krankheit tritt mit einer Wahrscheinlichkeit von 1:30000 Neugeborenen auf und manifestiert sich meist nach der Geburt. Durch das defekte Enzym OTC (EC 2.1.3.3) kann die Umwandlung von Ornithin zu Citrullin nicht oder nicht ausreichend stattfinden (siehe Abb. 1). Es kommt zu einer Anreichung von Ammoniak im Körper, wodurch schwere Symptome hervorgerufen werden können. Betroffene benötigen eine streng eiweißarme Diät.
Heute sind über eine Vielzahl angeborener Stoffwechselerkrankungen bereits viele Details bekannt, aber aktuelle Forschungsarbeiten, insbesondere auf dem Gebiet der Molekularbiologie, liefern ständig neue Erkenntnisse, die für die Behandlung dieser Erkrankungen neue Perspektiven eröffnen. Moderne Verfahren wie die gentechnologische Herstellung von Enzymen [2] oder die somatische Gentherapie [3,4,5] sind Beispiele für Therapieansätze, die für den Patienten möglicherweise eine bessere Erfolgsbilanz aufweisen als das mit den heute üblichen Therapienformen (Diät, Dialyse u.a.) erreichbar ist.
An der Fakultät für Informatik der Otto-von-Guericke-Universität Magdeburg wurde ein Informationssystem mit dem Namen MDDB - Metabolic Diseases Database – entwickelt [6]. Diese Datenbank konzentriert sich auf das genetische, medizinische und biochemische Wissen über angeborene Stoffwechselerkrankungen, wobei der Schwerpunkt auf Erkrankungen des Harnstoffzyklus liegt.
Das System MDDB wurde anfangs als Borland Interbase Datenbank mit einer mit Borland Delphi kreierten Nutzeroberfläche (GUI) realisiert. Dieses Konzept ermöglicht eine zentrale Dateneingabe und Weitergabe an den Nutzer auf CD-ROM und den Betrieb auf einem „standalone“ Rechner, beispielsweise einem Notebook ohne Internetverbindung. Der Nutzer kann die Daten einsehen und auch ergänzen. Ein Nachteil dieses Konzeptes ist die dezentrale Datenhaltung, denn Änderungen am Datenbestand sind nur für den Nutzer zugänglich, der diese Änderung durchgeführt hat. Aufgrund der rasant ansteigenden Zahl der mit dem Internet verbundenen Rechner erschien zu einem späteren Zeitpunkt des Projektes der Einsatz einer Client-Server-Datenbank mit Zugriff über das Internet als vorteilhafter, da bei einem derartigen System die Eingabe von Daten dezentral und von allen authorisierten Nutzer durchgeführt werden kann, während die Datenspeicherung aber zentral auf einem Server erfolgt. Auf diese Weise ist eine hohe Aktualität des Datenbestandes zu erreichen und für alle Nutzer zugänglich zu machen. Der Datenbestand der MDDB wurde auf eine Oracle-Datenbank portiert und erweitert. Die Nutzeroberfläche wurde mit JAVA realisiert, wobei der Datenzugriff über JDBC erfolgt. Das neue System wird als MD-Cave bezeichnet.
Die Daten in der MD-Cave (ebenso wie in der MDDB) gliedern sich in die vier Hauptkategorien: Krankheiten (Name, Häufigkeit, Anzahl der Fälle, Laborwerte, Prognose, Therapie usw.), Gene (Genebezeichnung, Genvarianten, Genregulation usw.), Enzyme (Name, Synonyme, EC-Nummer usw.) und Stoffwechselwege. Die Kategorie Stoffwechselwege zeigt den Stoffwechselweg (Metabolic Pathway), in dem die durch das defekte Enzym katalysierte biochemische Reaktion auftritt. Für viele Reaktionen existieren alternative Stoffwechselwege, die allerdings meist über mehrere Zwischenreaktionen laufen und eine weitaus geringere Reaktionsgeschwindigkeit aufweisen. Hier gibt es einen Ansatzpunkt für die Pharmaforschung, um mit neuen Wirkstoffen alternative Stoffwechselwege zu beschleunigen, indem die an den alternativen Stoffwechselwegen beteiligten Enzyme durch Agonisten aktiviert werden. Die MD-Cave kann alternative Stoffwechselwege berechnen und graphisch darstellen.
In der MD-Cave sind zwar auch Medikamente zur Behandlung der angeborenen Stoffwechselkrankheiten abgespeichert, zur Wirkungsweise der Medikamente bekommt man jedoch keine weiteren Informationen. An der Fakultät für Informatik der Otto-von-Guericke-Universität Magdeburg wird deshalb eine Wissensbasis für medikamentöse Wirkstoffe entwickelt. Ziel ist es, zwischen verschiedenen Wirkmechanismen zu unterscheiden und die jeweiligen Agenzien diesen zuzuordnen. Es soll dargestellt werden, wo im Stoffwechselweg die Medikamente eingreifen. Dadurch kann die Möglichkeit zur Aktivierung alternativer Stoffwechselwege zur Umgehung defekter Stoffwechselpfade sichtbar werden. Dabei sollen nicht nur klassische Medikamente erfaßt werden, sondern auch neuere Therapieverfahren, die sich teilweise noch im Versuchsstadium befinden, wie beispielsweise die Anwendung gentechnisch hergestellter Proteine oder die somatische Gentherapie. Ein wesentlicher Unterschied zu den klassischen Medikamenten, deren Wirkstoffe meist kleinere organische Moleküle (Liganden) sind, die an biologische Makromoleküle binden und dabei irgendeine Reaktion auslösen oder verhindern (inhibitieren), ist, daß die neueren Wirkstoffformen ursächlicher wirken und deshalb gezielter bestimmte Zellen erreichen müssen. Aufgrund ihrer Größe und Beschaffenheit können diese Moleküle ihr Target nur schwer erreichen, weil sie beispielsweise die Zellmembran nicht durchdringen können. Die Applikation ist deshalb schwieriger und bedarf weiterer Forschung.
Die Wirkstoffdatenbank MDDrugDB basiert ebenso wie die MD-Cave auf einer ORACLE-Datenbank, wobei die Nutzeroberfläche in JAVA programmiert werden soll. Diese Datenbank ist zwar eigenständig, aber MD-Cave und Wirkstoffdatenbank sollen sich gegenseitig ergänzen.

Abb. 1: Prinzipieller Zusammenhang zwischen DNA, Stoffwechsel, Krankheit und Wirkstoffen bei OTC-Mangel
[1] Mehnert, H.: Stoffwechselkrankheiten, Stuttgart: Georg Thieme Verlag, 1990
[2] Zündorf, I., Dingermann, T.: Gentechnologie und Arzneimittel, Pharmazie in unserer Zeit 27 (1998) 5
[3] Blankenstein, T. (Ed.): Gene Therapy - Principles and Applications, Basel: Birkhäuser, 1999
[4] Lemoine, N. R. (Ed.): Understanding Gene Therapy, Bios Scientific Publishers, 1999
[5] Heisig, P.: DNA als Arzneistoff, Pharmazie in unserer Zeit 228 (1999) 3
[6] Hofestädt, R., Prüß, M., Scholz, U., Urban, H.: Molekulare Bioinformatik – Molekulare Informationssysteme zur Erkennung von angeborenen Stoffwechselerkrankungen, Preprint Nr. 9, Fakultät für Informatik, Otto-von-Guericke-Universität Magdeburg 1998
[7] Freier, A.; Hofestädt, R.; Lange, M.; Scholz, U.: Integration, Modellierung und Simulation metabolischer Wirknetze, Preprint Nr. 13, Fakultät für Informatik, Otto-von-Guericke-Universität Magdeburg, 1999